相关性热图、圈图、弦图(笔记)
用图表、思维导图辅助笔记,更直观 #生活乐趣# #阅读乐趣# #阅读笔记#
首先非常欢迎大家在我们公众号平台分享自己与生物信息学/生信技能树的故事或者生信笔记,学习心得体会!下面是一年前的马拉松课程学员的分享
生信技能树优秀学员笔记,代码主要来自生信技能树的直播课程,加上自己的理解和发挥。
数据准备:
如下所示,可以看到有多个样品,每个样品都有多个基因表达量,这个时候我们比较关心的是这些基因的表达量相关性(在多个样品),基因与基因之间有两两组合相关性:
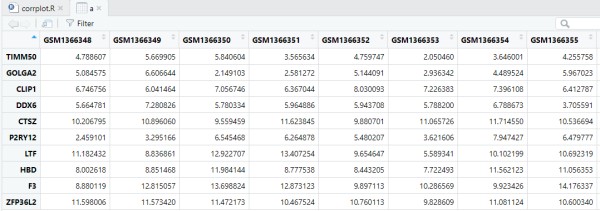
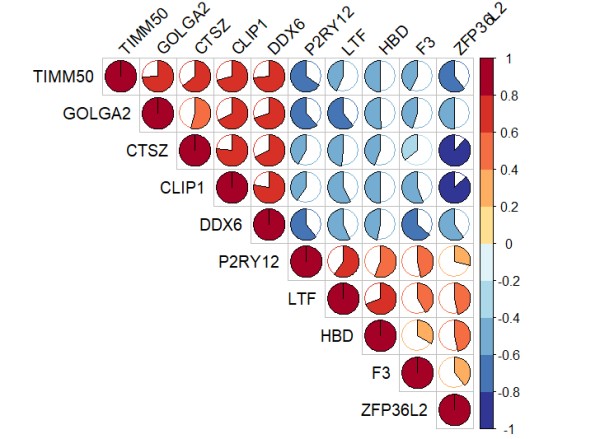
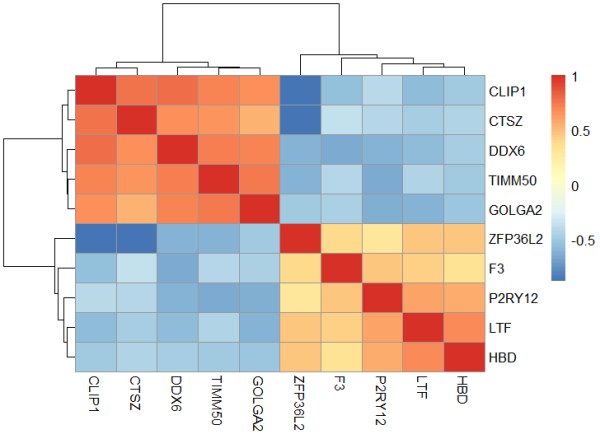
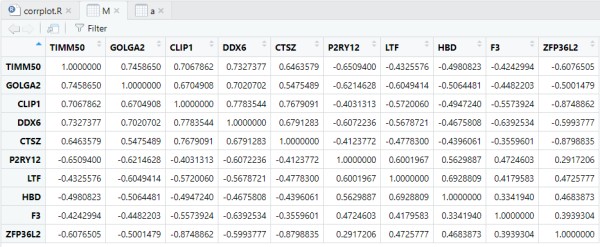
M: 很容易计算基因之间的相关性矩阵
代码语言:javascript
AI代码解释
复制
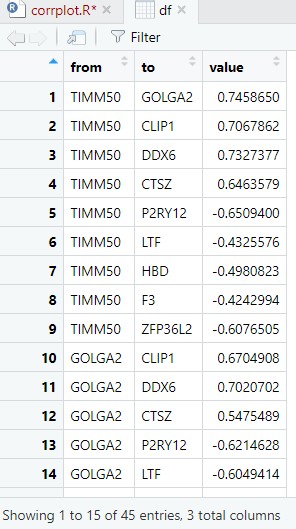
#感兴趣基因/样本的相关性图---- rm(list = ls()) load(file = "step1output.Rdata") load(file = "step4output.Rdata") load(file = 'step2output.Rdata') library(dplyr) library(ggplot2) library(pheatmap) library(corrplot) dat = deg[!duplicated(deg$symbol),] exp = exp[dat$probe_id,] rownames(exp) = dat$symbol g = c("TIMM50", "GOLGA2", "CLIP1", "DDX6", "CTSZ", "P2RY12", "LTF", "HBD", "F3", "ZFP36L2") a = exp[g,] M = cor(t(exp[g,])) #cor()函数可以对矩阵计算相关性(谁在列的位置就计算谁的相关性) #画基因之间的相关性,cor函数后面的矩阵exp[g,]要以基因为列名(转置一下) #画样本之间的相关性,cor函数后面的矩阵exp[g,]要以样本为列名(不要转置) #相关性热图 pheatmap(M) #相关性圆圈图 library(paletteer) my_color = rev(paletteer_d("RColorBrewer::RdYlBu")) my_color my_color = colorRampPalette(my_color)(10) #整成10个色阶 # library(RColorBrewer) # my_color=rev(brewer.pal(10, "RdYlBu")) corrplot(M, type="upper", method="pie", order="hclust", col=my_color, tl.col="black", #tl.pos = "d", tl.srt=45 ) #corrplot不能赋值,和其他R包拼图就比较费劲 library(cowplot) #比patchwork包更强的拼图包 cor_plot <- recordPlot() #cowplot包里的recordPlot函数可以把这个相关性图抠下来强行赋值 # 拼图(相关性图属于另外一个拼图体系) #load("pca_plot.Rdata") pdf("cor_plot.pdf", width = 10, height = 10) plot_grid(cor_plot) #plot_grid(pca_plot,cor_plot) #cowplot包里的plot_grid()函数允许跨绘图体系拼图 dev.off()
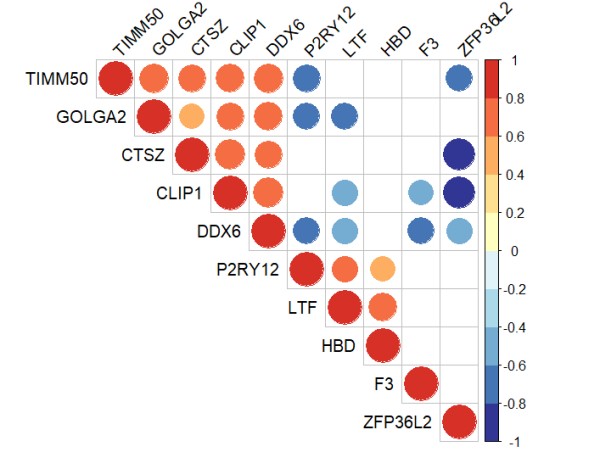
代码语言:javascript
AI代码解释
复制
## 圆圈视图 M = cor(t(a)) p.mat <- cor.mtest(t(a))$p library(paletteer) my_color = rev(paletteer_d("RColorBrewer::RdYlBu")[-1]) my_color = colorRampPalette(my_color)(10) png("plot/corrplot.png",height = 1000,width = 1000) corrplot(M, type="upper", order="hclust", col = my_color, p.mat = p.mat, sig.level = 0.01, insig = "blank", tl.col = "black", tl.srt=45) dev.off()
相关性弦图:连线就代表了基因间表达值的相关性信息,红色代表正相关,绿色代表负相关,颜色越深或连线越粗代表相关强度越高。 一行是一个连接关系,第一列和第二列是要连接的两边,第三列是连接的强度。
代码语言:javascript
AI代码解释
复制
## 相关性弦图 library(circlize) library(tidyr) library(tibble) library(ComplexHeatmap) mat = M df = mat %>% as.data.frame() %>% rownames_to_column(var = "from") %>% pivot_longer(names_to = "to", values_to = "value", cols = 2:(nrow(mat)+1) #有10个基因,这里就是11 ) df = df[df$from != df$to,] df = df[!duplicated(df$value),] #自定义边的颜色 library(RColorBrewer) col_sample = c(brewer.pal(n = 12,name = "Set3") #brewer.pal(n = 8,name = "Set2") ) col_sample border_color <- sample(col_sample,nrow(mat)) #border_color = c("#66C2A5", "#FC8D62", "#8DA0CB", "#E78AC3") #根据相关性大小展示连线的颜色范围:红 白 绿 col = colorRamp2(c(-1, 0, 1), c('green', 'white', 'red'), transparency = 0.5) # range(mat) # col <- colorRamp2(range(mat), c("#FA0A0A", "#FFFFFF")) # ComplexHeatmapt添加图例 lgd = Legend(col_fun = col, title = "foo") ComplexHeatmap:::width(lgd) ComplexHeatmap:::height(lgd) pushViewport(viewport(width = 0.9, height = 0.9)) grid.rect() # border draw(lgd, x = unit(1, "cm"), y = unit(1, "cm"), just = c("left", "bottom")) # draw(lgd, x = unit(0.5, "npc"), y = unit(0.5, "npc")) # draw(lgd, x = unit(1, "npc"), y = unit(1, "npc"), just = c("right", "top")) popViewport() chordDiagram(x = df, directional = 0, #表示线条的方向,0代表没有方向,1代表正向,-1代表反向,2代表双向 grid.col = border_color, #圈的颜色 col = col, #弦的颜色 annotationTrack = c('grid', 'name', 'axis'), #绘制外周圆弧区,显示名称和刻度轴 annotationTrackHeight = c(0.05, 0.1),#外面一圈的宽度 transparency = 0.25#线条的透明度 )
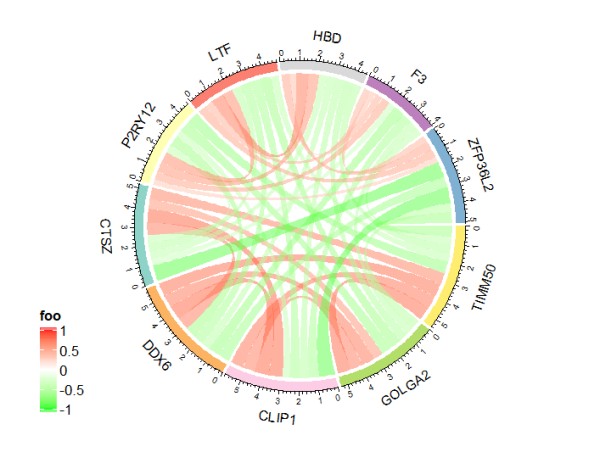
以下是小洁老师看到我的需求后发来的资料投喂: 弦图的边距问题: R circlize - 绘制边距和绘制区域- circlize - 码客 (oomake.com) 弦图函数的书: Chapter 16 A complex example of Chord diagram | Circular Visualization in R (jokergoo.github.io) 弦图图例用complexheatmap实现: Chapter 5 Legends | ComplexHeatmap Complete Reference (jokergoo.github.io
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-02-19,如有侵权请联系 [email protected] 删除
网址:相关性热图、圈图、弦图(笔记) https://c.klqsh.com/news/view/324051
相关内容
从海洋图鉴到美食探索:《海错图笔记》的深度之旅网络流行热词趣味性爆料热度娱乐圈人物手绘公众号封面首图
娱乐圈八卦热点独播趣味封面首图
App热图
Win11笔记本桌面无“此电脑”图标?实用解决指南
朋友圈图片设计
朋友圈背景图片带字励志,简单又个性!
时尚圈圈花纹边框矢量图图片
思维导图写日记的乐趣
励志和AI相关图片

